Index
AG Alexander Kaier
Ernäherungssicherung, Verbrauchervorlieben und die unmittelbare Bedrohung durch den globalen Klimawandel stellen große Ansprüche an Züchter, schnelle und effiziente Wege zu finden für die Entwicklung neuer Nutzpflanzensorten. Dementsprechend sind ein tiefes Verständnis der Toleranz gegenüber abiotischen Stressfaktoren von unschätzbarem Wert für die Idetifizierung neuer Ansäte für die gezielte Anpassung von Nutzpflanzen.
Der Fokus unserer Forschung liegt auf der Analyse diverser Nutzpflanzen, von denen manche nur lokal angebaut werden, andere hingegen zur Sicherung der weltweiten Narhrungsversorgung beitragen. Das Ziel ist dabei die Identifizierung genomischer und transkriptioneller Variationen, deren Nutzung zu einer erhöhten Toleranz gegenüber verschiedenen abiotischen Stressoren führen können. Dabei bedienen wir uns biostatistischer und bioinformatischer Methoden aus den Bereichen der quantitativen Genetik und Populationsgenetik. Ebenso benutzen wir modernste Techniken aus dem Bereich des maschinellen Lernens. Gemeinsam helfen uns diese Methoden dabei, komplexe Datensätze aus den Bereichen der Genomik, Transkriptomik, Methylomik, Metabolomik und Phenomik zu analysieren und zu integrieren. Die Interpretation der Ergebnisse helfen dabei, das vielschichtige Interaktionsnetz biologischer Systeme sowie die genetische Grundlage komplexer Merkmale besser zu verstehen. Das gewonnene Wissen trägt ebenso dazu bei neue Ziele für die molekulare Züchtung zu identifizieren.
Derzeit beschäftigen wir uns überwiegend mit die Analyse von Multi-Omik Datensätzen aus der Kartoffel (Solanum tuberosum). Nach Reis und Weizen stellt sie die wichtigste Nutzpflanze zur Erhaltung der Nahrungsversorgung dar. Die modernen Zuchtsorten haben ihren Urpsrung in den Hochlagen der Anden zwischenBolivien und Peru. Ihre Domestizierug liegt ca. 8.000 Jahre zurück. Die kultivierte Kartoffel wurde in den 70er Jahren des 16. Jahrhunderts in Europa eingeführt. Heutzutage wird die Kartoffel weltweit in Breitengraden zwischen 65°N und 50°S und bis auf 4.000 m Höhe angebaut. Dies demonstriert die erstaunliche Anpassungsfähigkeit der Kartoffel and unterschiedliche Umweltbedingungen. Trotz ihrer Anpassungsfähigkeit zeigt die Pflanze eine hohe Sensititvät gegenüber manchen abiotischen Stressoren, unter ihnen Trockenheit und Hitze. Vor allem hohe Temperaturen (über 20°C) haben einen erheblichen negativen Einfluss auf die Knollenproduktion.
Auf Grund des sich rasch verändernden Klimas, einer wachsenden Weltbevölkerung und der Notwendigkeit der Nahrungsproduktion unter suboptimalen Bedingungen, arbeiten wir mit nationalen und internationalen Partnern im derzeitigen Hauptprojekt. Für Details zum Hauptprojekt kann der Doktorand/die Doktorandin oder der Arbeitsgruppenleiter (Alexander Kaier) kontaktiert werden.
- Epigenetik der Hitzetoleranz in Kartoffel (Darren Yeo)
Im Laufe des vergangenen Jahrzehnts hat sich zunehmend abgezeichnet, dass agronomisch wichtige Merkmale in Nutzpflanzen durch einwirkende Umwelteinflüsse untrennbar mit ihrer epigenetischen Regulation verbunden sind. Als epigenetische Regulation wird die Steuerung der Genexpression durch Genommodifikation bezeichnet, die nicht die Genomsequenz verändern. Eine solche epigenetische Modifikation ist die Methylierung und Demethylierung des Cytosins. Methylierungsmuster können unterschiedlich Ausprägungen aufweisen zwischen Genotypen, einzelnen Pflanzen oder sogar zwischen verschiedenen Gewebetypen. Auch Umwelteinflüsse wie Hitzestress können zu differenzieller Methylierung führen, möglicherweise auch in der Kartoffelpflanze. Um diese Prozesse besser zu verstehen, untersuchen wir die genomweiten Methylierungsmuster von Kartoffelsorten, die ein kontrastierenden Verhalten im Bezug auf ihre Hitzetoleranz aufweisen. Unterschiede in der Methylierung des Genoms könnten Aufschluss geben über Mechanismen, die die Hitzetoleranz dieser wichtigen Nutzpflanze beeinflussen.
„CASS Transformation and Research Lab“ – AG Christian Lamm
Als Teil des CASS-Projekts arbeiten wir im CASS Transformation and Research Lab an der Entwicklung von in vitro-Methoden, um die genetische Transformation landwirtschaftlich nutzbarer Cassava-Genotypen zu ermöglichen, und stellen für unsere Projektpartner routinemäßig gentechnisch veränderte Cassava-Pflanzen her.
Die grüne Gentechnik ist für die moderne Pflanzenforschung und -züchtung unverzichtbar. Sie kann beispielsweise genutzt werden, um die molekularen Funktionen von Enzymen und Proteinen aufzuklären, bestehende Stoffwechselwege zu verbessern oder gänzlich neue einzuführen. Auch moderne Genomeditierungsverfahren wie das CRISPR/Cas-System sind i.d.R. auf klassische gentechnische Methoden angewiesen.
Die langwierigen Verfahren erfordern jedoch eine präzise Abstimmung auf die Versuchspflanze. Tatsächlich lassen sich selbst innerhalb einer Art meist nur wenige Kultivare effizient transformieren, ein Nachteil vor allem bei der gezielten Veränderung von Nutzpflanzen: Zugängliche Kultivare entsprechen nicht notwendigerweise den landwirtschaftlichen Bedürfnissen, zum Beispiel aufgrund geringerer Erträge oder höherer Anfälligkeit für Pathogene oder widrige Umweltbedingungen. Insbesondere das Aufkommen von Genomeditierungsverfahren hat daher dazu geführt, dass in der Forschung Wege gesucht werden, landwirtschaftlich nutzbare Sorten verschiedenster Kulturpflanzen zu transformieren.
In Zeiten des globalen Klimawandels und steigender Bevölkerungszahlen könnte die grüne Gentechnik so, Hand in Hand mit modernen Züchtungsverfahren, einen entscheidenden Beitrag zur dringend notwendigen Sicherstellung der weltweiten Nahrungsmittelsicherheit leisten.
Im Rahmen des CASS-Projekts beschäftigen wir uns in diesem Zusammenhang mit der tropischen Nutzpflanze Cassava, die vor allem von Kleinbauern in Sub-Sahara Afrika wegen ihrer stärkehaltigen Speicherwurzeln angebaut wird. Im internationalen Vergleich werden hier jedoch drastisch niedrigere Erträge erzielt, unter anderem wegen mangelnder Verfügbarkeit von Düngemitteln, Bioziden und modernen landwirtschaftlichen Geräten. Eine biotechnologische Anpassung zur Ertragsstabilisierung bei niedrigem Input ist daher unser erklärtes Ziel.
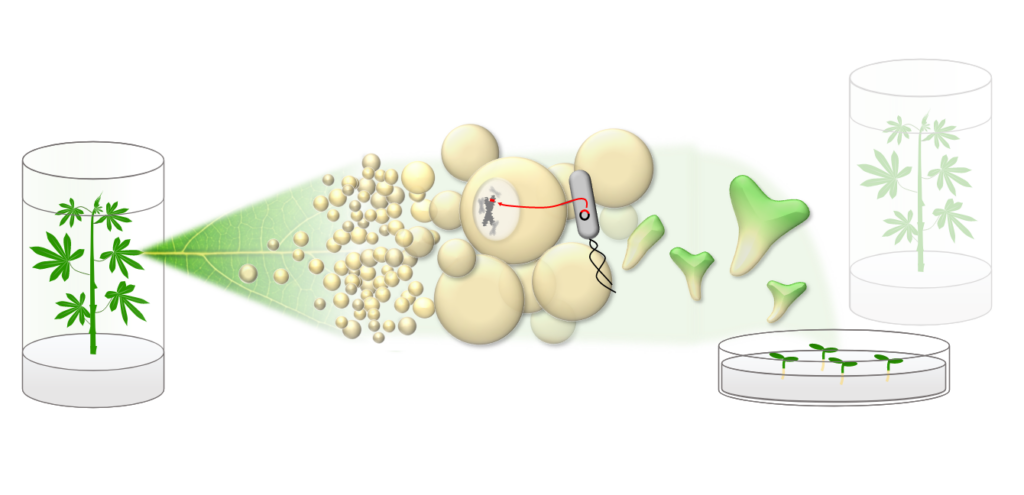
Abbildung 1: Schematische Darstellung der genetischen Transformation von Cassava: Blattexplantate werden genutzt, um eine Kalluskultur anzulegen. In die Zellen lässt sich durch Agrobacterium-vermittelte Transformation gezielt DNA einschleusen. Durch Änderung des Nährmediums durchlaufen die Zellen eine Embryogenese und können zu vollwertigen, transgenen Pflanzen regeneriert werden.

Im CASS Transformation and Research Lab beschäftigen wir uns mit der Weiterentwicklung der genetischen Transformation von Cassava, um landwirtschaftlich relevante Genotypen gentechnisch verändern zu können. Wir untersuchen dabei unter anderem den Nutzen chemischer Medienzusätze, die durch verschiedene Wirkmechanismen, z.B. auf epigenetischer Ebene, das Entwicklungsprogramm der pflanzlichen Zellen verändern und so eine Transformation und/oder Regeneration erleichtern bzw. ermöglichen sollen.
Zudem arbeiten wir mit genetischen Elementen, die gleichermaßen den Transformations- und Regenerationsprozess unterstützen. So kann die Expression bestimmter Entwicklungsregulatoren genutzt werden, bisher nicht transformierbare Kultivare gentechnisch zu verändern – ein Prinzip, dass andere Arbeitsgruppen bereits erfolgreich in Nutzpflanzen wie Mais einsetzen.

Neben diesen Forschungsfragen führen wir auch Routine-Transformationen des Cassava Modellgenotyps TMS60444 für unsere Projektpartner durch (Abbildung 1).
Dabei übernehmen wir den gesamten Arbeitsablauf: Cassava Pflanzen werden unter sterilen Bedingungen angezogen und mit Phytohormonen behandelt, um dedifferenziertes Kallusgewebe zu erzeugen. Ein geeignetes, durch molekulare Klonierung erzeugtes Transformationsplasmid wird durch Elektroporation in einen Laborstamm des Bodenbakteriums Agrobacterium tumefaciens eingeschleust.
Das Bakterium ist bereits natürlicherweise in der Lage, Erbgut in pflanzliche Zellen einzubringen, und durch Kultivierung mit dem Kallusgewebe kann so ein gezielter Transfer der gewünschten DNA erfolgen.
Durch Änderung in der Medienzusammensetzung können schließlich wieder vollwertige Pflanzen regeneriert werden, die das gewünschte genetische Material in das eigene Genom integriert haben (Abbildung 2).
Nach molekularer Bestätigung der genomischen Integration werden die Pflanzen schließlich vegetativ vermehrt und können weiter analysiert werden (Abbildung 3, 4).

„Cassava Source-Sink“ – AG Wolfgang Zierer

Unser Team strebt danach den Ertrag von Cassava zu erhöhen. Cassava ist eine tropische Nutzpflanze die weltweit mehr als 800 Millionen Menschen mit Nahrung versorgt. Besonders in Sub-Sahara Afrika besitzt die Pflanze eine enorme Bedeutung für die Nahrungssicherheit der lokalen Kleinbauern.
Der 3-4 Meter hohe Strauch bringt unter guten Bedingungen zahlreiche stärkehaltige Speicherwurzeln hervor. Die Wurzeln (Abb. 1) und die Stärke in diesen Wurzeln bilden die Basis für vielfältige Nahrungsmittel.

So werden die Wurzeln beispielsweise gekocht oder frittiert gegessen, oder die Stärke in den Wurzeln wird zu Produkten wie dem afrikanische Gari, eine Art Brei, verarbeitet.
Im Gegensatz zu bereits sehr ertragsoptimierten Nutzpflanzen wie z.B. Weizen oder Mais, sind die Erträge von Cassava oft noch niedrig. Eine nachhaltige Ertragssteigerung würde daher die Nahrungssicherheit von Millionen Menschen verbessern. Deshalb arbeiten wir an der biotechnologischen Verbesserung des Cassava Ertrags.
Leider ist dieses Ziel noch nicht zum Greifen nahe und eine biotechnologische Verbesserung bedingt ein tiefgreifendes Verständnis der Pflanze. Unsere Arbeit beinhaltet deshalb auch sehr viel Grundlagenforschung um die physiologischen und biochemischen Prozesse dieser spannenden Pflanze besser zu verstehen.

Wir forschen vor allem an der Regulation des Kohlenstoffwechsels in autotrophen- und heterotrophen Pflanzengeweben, dem Assimilat Transport, sowie der pflanzlichen Entwicklung. Innerhalb dieser Prozesse versuchen wir Gene mit positiven Auswirkung auf den Ertrag zu identifizieren und sie eventuell miteinander zu kombinieren um einen noch stärkeren Effekt zu erzielen (Abb. 3).

Wir testen die Gene mit Hilfe von transgenen Cassavapflanzen, die wir entweder selbst oder mit Hilfe unserer Kooperationspartner an der ETH Zürich erstellen. Dabei kommen ausgefeilte Gewebekulturprotokolle zum Einsatz die aus de-differenzierten Zellen wieder ganze Pflanzen regenerieren (Abb. 4).
Die von uns veränderten Cassavapflanzen werden zunächst in unseren Gewächshäusern mit verschiedensten Methoden untersucht. Pflanzen die besser wachsen als ihr jeweiliger genomischer Hintergrund sind dabei natürlich besonders interessant (Abb. 5).

Neben der Erfassung von Wachstumsparametern und physiologischer Studien wie z.B. Photosynthese Messungen (Abb. 6), kommen bei uns alle gängigen Techniken molekularer Pflanzenforschung wie z.B. Transkriptom- und Proteomanalysen (Abb. 7) regelmäßig zum Einsatz.
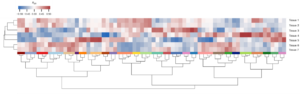
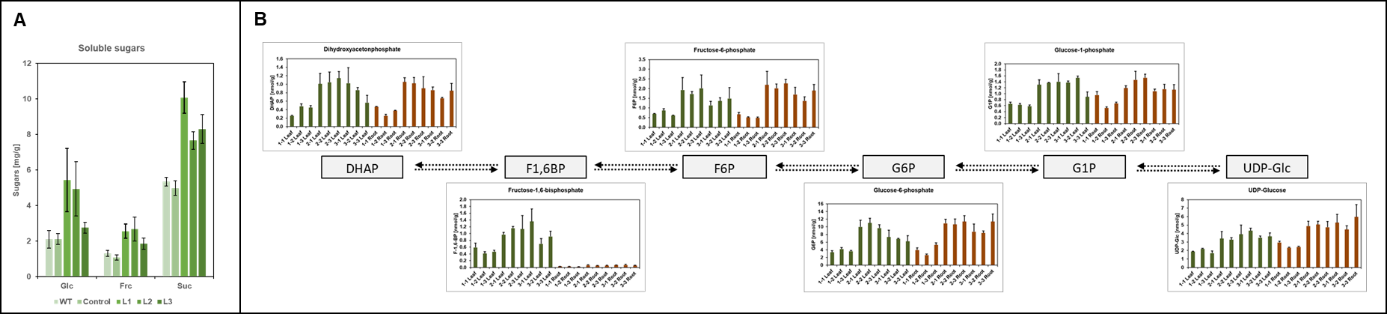
Auch zahlreiche Metabolit Messungen gehören zu unserem Standardrepertoir. So gewähren uns z.B. enzymatische Zuckermessungen und Massenspektrometrie-basierte Methoden Einblicke in den pflanzlichen Primärmetabolismus (Abb. 8).
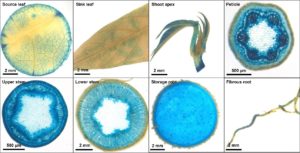
Zusätzlich zur Analyse von ertragsrelevanten Genen arbeiten wir auch an der Entwicklung von besseren Werkzeugen zur Erzeugung transgener Pflanzen wie z.B. verbesserte Transformationsprotokolle oder die Identifizierung gewebespezifischer Promotoren (Abb. 9).
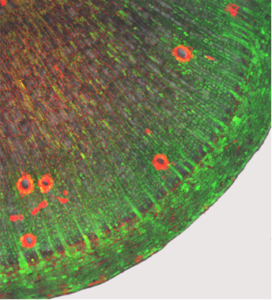
Desweitern führen wir auch verschiedene Studien an Cassava Wildtyp (WT) Pflanzen und verschiedenen Zuchtsorten durch um grundlegende physiologische Prozesse der Cassavapflanze wie z.B. die Speicherwurzelbildung oder den Assimilat Transport besser zu verstehen.
So haben wir zum Beispiel mit Beladungsexperimenten mit fluoreszierenden Substanzen die zelluläre Konnektivität von Cassava Stämmen und Speicherwurzeln aufgeklärt (Abb. 10).
Unsere Arbeitsgruppe ist in das Cassava Source-Sink (www.cass-research.org) Projekt (CASS) eingebettet und hat durch unsere zahlreichen Partner Zugriff auf ein größeren Netzwerk von pflanzenbiologischen Techniken und Expertisen (Abb. 11).
Wir pflegen enge Kooperationen mit:
- International Institute of Tropical Agriculture (IITA), Bioscience, Ibadan, Nigeria
- National Root Crops Research Institute (NRCRI), Umudike, Nigeria
- Chung-Hsing University (NCHU), Advanced Biotechnology Center, Taichung, Taiwan
- Sainsbury Laboratory Cambridge University (SLCU), Cambridge, United Kingdom

Abb. 12: Verschiedene Phasen der Feldtestung an der NCHU Taichung, Taiwan - Boyce Thompson Institute, Plant Research (BTI), Ithaca NY, USA
- Eidgenössische Technische Hochschule (ETH), Biochemie, Zürich, Schweiz.
- Technische Universität Kaiserslautern, Lehrstuhl für Pflanzenphysiologie, Deutschland
- Forschungszentrum Jülich, Institut für Bio- und Geowissenschaften, Deutschland
- Max-Planck-Institute, Molekulare Pflanzenphysiologie, Deutschland
Mit Hilfe unserer Partner haben wir eine vollständige Pipeline von der Erstellung transgener Pflanzen, über die Labor- und Gewächshausanalyse, bis hin zur Feldtestung aufgesetzt (Abb. 12).
Versuchsfeld in Taiwan: 3 Monate alte Maniok-Pflanzen
Versuchsfeld in Taiwan: Kurz vor der Ernte

Unsere Feldversuche finden gemeinsam mit der NCHU Taichung, Taiwan und dem IITA Ibadan, Nigeria (Abb. 13) statt.
An beiden Standorten testen wir transgene Cassavapflanzen mit Änderungen im Source-Sink Stoffwechsel. Am IITA Ibadan wird außerdem Cassava auf konventionelle Art gezüchtet und wir arbeiten zusammen an zahlreichen Projekten zur Charakterisierung dieser Genotypen.
Unsere Arbeitsgruppe und das gesamte Cassava Source-Sink Projekt hoffen einen kleinen Beitrag zur Verbesserung der Nahrungssicherheit afrikanischer Kleinbauern leisten zu können. Wenn ihr euch auch für Cassava interessiert oder uns bei unserem Projekt unterstützen wollt, meldet euch gerne bei wolfgang.zierer@fau.de.
Bioanalytik – AG Jörg Hofmann
Die Bioanalytikgruppe betreibt eine Metabolomikplattform und eine Proteomikplattform zur qualitativen und quantitativen Analyse von Biomolekülen. Zentrale Projekte schließen gerichtetes und ungerichtetes Metabolit-Profiling ein. Zur Zeit besteht unsere Plattform aus folgenden Komponenten:
Metabolomik Plattform
LC/MS-MS System
- ABI/MDS-Sciex QTRAP-3200 (Massenspektrometer)
- Dionex ICS-3000 Ionenaustausch-HPLC (Metabolitanalyse)
- Dionex ULTIMATE-3000 HPLC (Metabolitanalyse)
Weitere Geräte
- Dionex SUMMIT P680 HPLC (Metabolitanalyse)
- Shimadzu GCMS-QP2010S mit GC-2010 (Fettsäureanalyse)
- Äkta Purifier HPLC (Proteinreinigung)
- Pharmacia P-800 FPLC (Niederdrucksystem)
- Packard Flow Scintillation Analyser (Radioaktive Moleküle)
- BIO-TEK ELISA Mikrotiterplatten-Fotometer (Visuell/Fluoreszenz)
- Goebel UVIKON-XL Spektrofotometer (UV/Vis)
Nutzung
Für die Verwendung der Analyseplattform ist nach vorheriger Absprache unter Beachtung der Nutzungsordnung (PDF) ein Probenformular per E-Mail anzufordern, auszufüllen und per E-Mail einzusenden an: joerg.hofmann (at) fau.de
Das Kernstück unserer LC-MS/MS-Analytikplattform ist ein vielseitig einsetzbares Hybrid-Massenspektrometer des Typs QTRAP-3200. Seine Einsatzstärke ergibt sich aus der Kombination eines dreifach-kaskadierten Massenfilters (Triple-Quadrupole) welches für höchste Selektivität sorgt, mit einer linearen Ionenfalle, welche umfangreiche Möglichkeiten zur Strukturaufklärung bietet. Das Dreifachmassenfilter in Kombination mit speziell gestalteten Schnittstellen der Ionenquelle (Stickstoffvorhang) erlaubt die Analyse relativ stark verunreinigter Proben. Zur Messung instabiler Moleküle lässt sich den Härtegrad der Ionisation beeinflussen, durch Auswahl zweier Ionenquellen ( Gas assisted Electro Spray Ionisation (ESI), Atmospheric pressure chemical ionisation (APCI) ) eine optimale Anpassung an verschiedene Substanzklassen erzielen. Die Ionenquelle erhält einen konstanten Eingangsstrom von Analyten nach Auftrennung in einer Ionenchromatografie-Anlage (ICS-3000) oder eines HPLC-Systems (ULTIMATE-3000).
Die inerte ICS-3000-Anlage eignet sich speziell zur Ionenaustauschchromatografie z.B. von Kohlehydraten und phosphorylierten Intermediaten. Sie erzeugt ein „reaktantfreies“ Laufmittel welches für Massenspektrometrie erforderlich ist und ist mit eigenen Detektoren ausgestattet (Leitfähigkeitsdetektor für z.B. phosphorylierte Intermediate, elektrochemischer Detektor für z.B. Zucker). In Verbindung mit dem QTRAP-Massenspektrometer können wir dieses System für ein gerichtetes Metabolit-Profiling mit hohem Dynamikumfang einer umfangreichen Palette von Verbindungen einsetzen.
Die Kopplung der ULTIMATE-3000-HPLC mit einem UV/Vis Photodiode Array Detector (PDA) ermöglicht eine 3D-Visualisierung von Fluoreszenzsignalen zur schnellen Identifikation und genauen Bestimmung von z.B. Pigmenten wie Carotinoiden oder Chlorophyll.
Ein weiteres HPLC-System (Dionex SUMMIT) ausgestattet mit einem Fluoreszenzdetektor erlaubt die sensitive Messung von Verbindungen wie Tocopherol und (derivatisierter) Aminosäuren mittels Reversed Phase- (RP-)Chromatografie.
Die GC/MS-Anlage (Shimadzu) setzen wir zur Bestimmung von stark hydrophoben Molekülen wie Fettsäuren ein.
Weitere Geräte erweitern unser Methodenspektrum: Simultananalysen grösserer Probenzahlen z.B. zur Zucker-Bestimmung können wir mit einem Mikrotiterplatten-Fotometer (96-well ELISA plate reader) mit Waschgerät und Fluoreszenzdetektor sowie zwei Uvikon UV/Vis-Spektrofotometern durchführen. Ein Flow Scintillation Analyser (Packard) ermöglicht uns radioaktiv markierte Verbindungen nach chromatografischer Auftrennung zu detektieren. Ferner steht uns ein gekühlter Äkta-Purifier (HPLC, General Electric) und eine Pharmacia-FPLC für die Proteinanalytik zur Verfügung.
Diese Ausrüstung erlaubt die parallele Untersuchung von Metaboliten in komplexen Gemischen und unterstützt Projekte zur Erforschung von Interaktionen zwischen Pflanzen und Mikroorganismen sowie des pflanzlichen Primär-Stoffwechsels ( AG S.Sonnewald, AG U.Sonnewald. Daneben werden auch Verbindungen zwischen Pflanzenzellen (Plasmodesmata) auf biochemischer und molekularer Ebene analysiert.
Proteomik Plattform
Innerhalb des Zentralprojekts Z1 im Sonderforschungsbereich SFB796 wurde 2014 ein Proteomik-Labor eingerichtet. Dies ermöglicht die Identifikation und Analyse von Proteinen und Proteinkomplexen inklusive ihrer post-translationalen Modifikationen, basierend auf hochauflösender Massenspektrometrie.

Zentrale Komponenten der Plattform sind die Systeme Ultimate 3000 nano-HPLC und Orbitrap Fusion Tribrid zur bioanalytischen Forschung im Bereich Proteomik. Optimierte Verfahren der Probenvorbereitung und Analyse ermöglichen uns in Aufreinigungsfraktionen, intakten Proteingemischen, SDS-PAGE Bands/Spots und immunopräzipitierten Proteinen (On/Off-Beads) die Identifikation von
- Interaktionspartnern
- PTMs
- Unterschieden auf Proteinebene (z.B. Wildtyp vs. Mutante, Transfektion, Differenzielle Expression, Stresseinfluss)
- Proteom/Sekretom Studien
Spezielle Projekte wie Biomarker-Erkennung, Quantifizierung (Labelled/Label-free) und Phosphoproteom-Analyse befinden sich in der Entwicklungsphase.
Betrieb und Management der Plattform: Jörg Hofmann
Für weitere Auskunft kontaktieren Sie: joerg.hofmann (at) fau.de , uwe.sonnewald (at) fau.de
Formular zur Einreichung von Proben:
PDF als Datei herunterladen, ausfüllen, in eine PDF-Datei ausdrucken und per E-Mail senden.
Klimaanpassungen von Kartoffeln – AG Sophia Sonnewald
Meine Forschungsgruppe interessiert sich hauptsächlich für die Regulation der „source-sink“- Wechselwirkungen während der Pflanzenentwicklung und durch widrige Umweltbedingungen wie z.B. Hitze und Trockenheit. Als Modellsystem verwenden wir hauptsächlich Kartoffelpflanzen. Die Kartoffel gehört zu den wichtigsten Kulturpflanzen der Welt. Ihre Knollen sind ein hervorragendes Grundnahrungsmittel, da sie reich an Stärke sind, wertvolle Mineralien, Vitamine und essentielle Aminosäuren enthalten. Kartoffelpflanzen sind jedoch empfindlich gegenüber Hitze und Trockenheit, und es wird erwartet, dass globale Klimaveränderungen die Ertragsstabilität und die Qualität der Knollen stark beeinträchtigen.
Unsere aktuelle Forschung konzentriert sich auf zwei Hauptthemen:
- Die Reaktionen von Kartoffelpflanzen auf Hitze und Dürre besser zu verstehen, um sie für die Herausforderungen des Klimawandels zu verbessern
- Die molekulare Analyse der Kartoffelknollenentwicklung
Verbesserte Kartoffelpflanzen für zukünftige Herausforderungen des Klimawandels
Erhöhte Temperaturen beeinflussen viele physiologische Prozesse und Entwicklungsprozesse in Kartoffelpflanzen. Dazu gehören u.a. die verminderte Produktion von Assimilaten durch die Photosynthese und die negativen Auswirkungen auf die Knollenentwicklung, die Stärkeakkumulation und die Qualität von Kartoffelknollen.
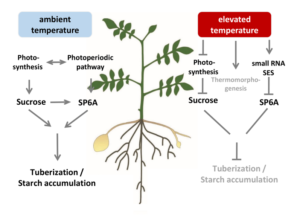
Ein wichtiger Regulator dieser Prozesse ist das „Flowering Locus T“-Homolog SP6A, welches ein Schlüsselgen für die Knollenbildung ist. Die Genexpression von SP6A wird durch Hitze verringert, was mit verringerter Knollenbildung und -wachstum einhergeht. In unseren Arbeiten haben wir eine kleine RNA (genannt SES) entdeckt, die durch erhöhte Temperaturen stark induziert wird, an das SP6A Transkript bindet und dieses post-transkriptionell abbaut (Lehretz et al. 2019; siehe Abbildung 1).
Abbildung 1: Schematische Darstellung der Auswirkungen erhöhter Temperaturen auf den Stoffwechsel und die Entwicklung von Kartoffelpflanzen. Die photosynthetische Kohlenstoffproduktion und -allokation wird gehemmt. Die Transkriptakkumulation des Schlüsselgens für die Knollenbildung SP6A wird durch eine hitzeinduzierte kleine RNA (namens SES) verringert. Zusammen mit der Induktion von Thermomorphogenese und anderen Effekten führt bei Hitze zu einer Hemmung der Knollenentwicklung und/oder einer verringerten Stärkeakkumulation.
- Gegenwärtig arbeiten wir daran (DFG HotNet) das regulatorische Netzwerk, welches unter Hitzestress in Kartoffelpflanzen wirkt, weiter zu entschlüsseln, indem wir physiologische und biochemische mit molekularen und genetischen Ansätzen kombinieren. Wir nutzen die genetische Variabilität (Abbildung 2), um Zielgene aufzuklären, da wir denken, dass innerhalb der verfügbaren genetischen Ressourcen ein großes Potenzial zur Erhöhung der Ertragsstabilität besteht. Die Kartoffel ist eine komplexe, hochgradig heterozygote, tetraploide Nutzpflanze, die einfache genetische Ansätze erschwert. Zusammen mit der Bioinformatik-Gruppe von Dr. Stephan Reinert führen wir eine detaillierte Phänotypisierung verschiedener tetraploider Sorten durch und möchten mit einem mittels eines GWAS-Ansatzes und durch Transkript-Profiling-Experimente neue, interessante Kandidatengene identifizieren.
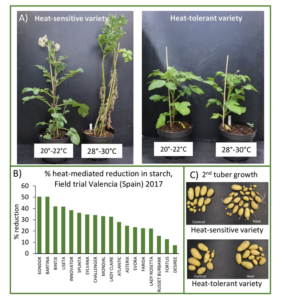
Abbildung 2: Reaktionen auf Hitzestress unterscheiden sich je nach Kartoffel-Genotyp. Erhöhte Temperaturen induzieren eine stärkere Thermomorphogenese (A), eine dramatischere Abnahme des Knollenstärkegehalts (B) und ein häufigeres Auftreten von 2. Knollenwachstum (C) bei anfälligen Sorten als bei toleranten.
- Im Rahmen des von der EU finanzierten Horizon 2020-Projekts Adapt wollen wir ein besseres Verständnis der Reaktionen von Kartoffelpflanzen auf kombinierten Stress, insbesondere auf Hitze, Trockenheit und Staunässe, gewinnen. Adapt ist ein Forschungskonsortium von 17 Partnern aus führenden akademischen Forschungseinrichtungen, Kartoffelzüchtern, einem gemeinnützigen EU-Verband, einer Regierungsbehörde und einem Entwickler von bildgebenden Hochdurchsatz-Technologien. Ziel ist es, neue Züchtungsansätze und Kartoffelsorten zu identifizieren, die eine Anpassung an sich verändernde Umweltwachstumsbedingungen in der Zukunft ermöglichen. Unter anderem werden wir die Rolle von SP6A und der kleinen regulatorischen RNA SES weiter analysieren. Weitere Informationen und Updates finden Sie auf der offiziellen Webseite des Projekts (adapt.univie.ac.at) und auf dem Twitter-Account (@eu_Adapt).
- Veränderte Umweltbedingungen führen auch zu einem reduzierten Knollenstärkegehalt und können Zwiewuchs bei Knollen verursachen (Abb. 2b, c). Beide Merkmale führen zu einem Qualitätsverlust der Knollen und erschweren deren Verwendung. Daher versucht meine Gruppe, die zugrundeliegenden molekularen Mechanismen aufzuklären.
Molekulare Analyse der Kartoffelknollenentwicklung
- Die Entwicklung von Kartoffelknollen hängt stark von Stoffwechsel- und Entwicklungssignalen der Blätter ab. Der photoperiodische Weg mit Constans und SP6A als Schlüsselregulatoren spielt eine wichtige Rolle dabei und steuert die Knollenbildung und deren Wachstum. Jüngste Arbeiten deckten auf, dass SP6A mit Saccharose-Efflux-Transportern wie Sweet11b interagiert und dadurch die Assimilat-Translokation zu sich entwickelnden Knollen fördern kann. Auch wenn unser Wissen in letzter Zeit zugenommen hat, gibt es noch viele offene Fragen zur Wirkungsweise und den endogenen und umweltbedingten Regulatoren. Wir führen derzeit molekulare und biochemische Analysen durch, um (i) die Rolle des Blaulichtrezeptors FKF1 bei der Regulierung der Knollenentwicklung zu entschlüsseln und (ii) um ein besseres Verständnis dafür zu erhalten, wie SP6A die Assimilationsverteilung und die „Source-Sink“- Beziehungen verändert.
Pflanzen-Biochemie und -Biotechnologie – AG Uwe Sonnewald
Die AG beschäftigt sich mit anwendungsorientierter Grundlagenforschung in den Bereichen Pflanzenwachstum und -entwicklung sowie mit Aspekten der Synthetischen Biologie. Pflanzenwachstum wird durch interne und externe Faktoren reguliert, wobei prognostizierte Klimaveränderungen eine große Herausforderung für die Landwirtschaft darstellen. In diesem Zusammenhang untersuchen wir molekulare Hintergründe der Anpassung von Pflanzen an Hitze- und Dürreperioden, um sie fit für den Klimawandel zu machen. Um die Produktivität von Nutzpflanzen wie Kartoffel oder Maniok zu erhöhen verfolgen wir einen biotechnologischen Ansatz zur Verbesserung der Assimilatproduktion, -verteilung und -nutzung. Im Bereich der Synthetischen Biologie versuchen wir durch gezielte Protein-Protein Wechselwirkungen den pflanzlichen Stoffwechsel so umzuprogrammieren, dass er besser an zukünftige Klimabedingungen angepasst ist. Ein Kernstück dieser Arbeiten stellen intermolekulare, bruchfeste und spezifische Proteinverknüpfungen dar ( doi: 10.1186/s13007-020-00663-9 ; doi: 10.1371/journal.pone.0179740 ).
Anpassung an den Klimawandel:
Gemäß unabhängiger Klimamodelle ist innerhalb der nächsten Dekaden mit einer Umverteilung der jährlichen Niederschläge und einem Anstieg der Durchschnittstemperaturen in Deutschland zu rechnen. Es ist jedoch kaum erforscht, welche Anpassungsmechanismen Nutzpflanzen besonders effektiv gegen kombinierten Hitze- und Trockenstress schützen können. Bereits eine moderate Erhöhung der atmosphärischen Temperatur kann drastische Effekte auf die Ertragsbildung der Kartoffelpflanzen haben. Hierbei spielt die Beeinflussung der Bildung der Knollen eine wesentliche Rolle (doi.org/10.1146/annurev-arplant-080720-084456).
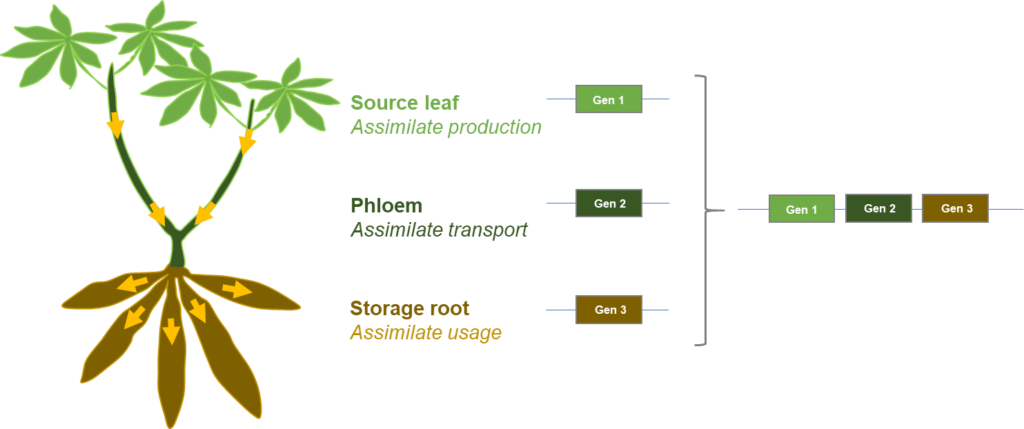
Verbesserung der Source-Sink Verhältnisse in Nutzpflanzen:
Der Ertrag von Nutzpflanzen wird maßgeblich durch das Wechselspiel zwischen Blättern und Speicherorganen bestimmt. Hierbei überführen die Blätter atmosphärisches Kohlendioxid in organische Kohlenstoffverbindungen (ein Prozess der Photosynthese genannt wird), wobei als „Abfallprodukt“ Sauerstoff entsteht. Da ausgewachsene Blätter tagsüber mehr Kohlenhydrate produzieren, als sie nachts verbrauchen, können sie den Überschuss anderen Organen zur Verfügung stellen. Man bezeichnet die Blätter als Source (Quellen) Organe. Die überschüssigen organischen Verbindungen werden von den Blättern über das Leitbahnsystem der Pflanze in verbrauchende oder speichernde Gewebe (Sink Organe) transportiert. Angekommen in heterotrophen Geweben, werden die Verbindungen entweder für das Wachstum (z.B. Wurzeln) oder die Speicherung (in Samen, Knollen, etc.) eingesetzt. Je mehr Assimilate zum Aufbau von Speichersubstanzen verwendet werden, desto größer ist der Ertrag. Daher richten sich die Interessen der Pflanzenzüchtung darauf, soviel Assimilate wie möglich in Speicherorgane einzulagern. Basierend auf einem zunehmenden Verständnis der Prozesse, die sowohl den Blattstoffwechsel als auch den Stoffwechsel min Speicherorganen betrifft wurden eine Reihe biotechnologischer Verfahren entwickelt, die Ertragssteigerungen versprechen (zusammengefasst in doi.org/10.1016/j.molp.2022.11.015). Das Verhältnis zwischen Gesamtbiomasse und erntebarer Biomasse bezeichnet man hierbei als Ernteindex. Im Rahmen zweier internationaler Forschungsprojekte ( www.photoboost.org ; cass-research.org ) versuchen wir gezielt das Wechselspiel zwischen Blatt und Speicherwurzel der Maniokpflanzen bzw. zwischen Blatt und Speicherknolle der Kartoffelpflanzen zu verändern, so dass höhere Erträge realisiert werden können. Hierbei liegt das Augenmerk auf der simultanen Verbesserung sowohl des Blattstoffwechsels (Source), des Transports (Phloem) als auch des Stoffwechsels der Speicherwurzeln/ -knollen (Sink). Die Ansätze des Maniokprojektes sind in der Veröffentlichung von Sonnewald et al., 2020 ( doi: 10.1111/tpj.14865 ) zusammengefasst. Übergeordnete Ansätze werden in Fernie et al., 2020 ( doi: 10.1038/s41477-020-0590-x ) beschrieben.