Biocomputing – AG Stephan Reinert
Lebensmittelsicherheit, Verbraucherwünsche, und die zunehmend Gefahren durch die globale Klimaveränderung motivieren Pflanzenzüchter rasch umsetzbare und effiziente Wege zu finden, neue Kultursorten zu entwickeln. Deshalb ist es von höchster Bedeutung ein genaues und tiefgehendes Wissen über die abiotische Stresstoleranz und den zugrundeliegenden genetischen Faktoren zu erhalten, um neue Ansätze zur Optimierung von Nutzpflanzen zu entwickeln.
Das Ziel unserer Forschung ist die Analyse verschiedener Nutzpflanzen, um Kandidatengene, allelische Variationen und genetische Marker zu entdecken, welche mit agronomisch wichtigen Merkmalen assoziiert sind. Mit diesem Wissen können wir gezielt Anbauerträge, Qualität und Nachhaltigkeit verbessern. Um dies zu erreichen, setzen wir moderne statistische und bioinformatische Methoden aus den Bereichen der quantitativen und Populationsgenetik ein. Mit Hilfe dieser Methoden sind wir in der Lage Datensätzen aus Genomik, Transkriptomik, Metabolomik und Phänomik zusammenzuführen, um eine ganzheitliche Analyse zu ermöglichen. Dies erlaubt uns, genregulatorische Netzwerke und die genetischen Komponenten komplexer Merkmale zu identifizieren und neue Ansätze für die molekulare Züchtung zu erarbeiten.
Momentan fokusiert sich unsere Forschung auf die Multi-Omics Analyse von zwei wichtigen stärkehaltigen Nutzpflanzen, Cassava (Manihot esculenta) und Kartoffel (Solanum tuberosum).
Cassava (Manihot esculenta) ist ein Strauchgewächs, welches stärkehaltige unterirdische Speicherwurzeln entwickelt. Cassava zählt als eine der wichtigsten Nutzpflanzen weltweit, besonders in den tropischen und subtropischen der Erde. Obwohl Cassava als eine der wichtigsten Grundnahrungsmittelpflanzen für mehr als 800 Millionen Menschen in den Amerikas, Afrika und Asien zählt, wird Cassave hauptsächlich von Kleinbauern angebaut. Aufgrund des begrenzten Zugangs zu modernen landwirtschaftlichen Geräten und Chemikalien, wie Landmaschinen, Dünger und Pestiziden sind Kleinbauern abhängig von Pflanzen, die selbst unter suboptimalen Bedingungen hohe Erträge erzielen. Aus diesem Grund fokussiert sich unsere Forschung, als Teil des Cassava Source-Sink (CASS)-Projektes, auf die Entwicklung resistenter sowie ertragsoptimierter Cassavasorten, welche wir subsaharischen Kleinbauern in Afrika zur Verfügung stellen wollen, um die Grundernährung sicherstellen zu können.
Kartoffel (Solanum tuberosum) zählt als eine der wichtigsten Nutzpflanzen weltweit und liegt lediglich hinter den Getreiden Reis und Weizen. Aufgrund des bisher kontinuierlichen globalen Ertragsanstiegs wird Kartoffel den essenziellen Grundnahrungsmittelpflanzen zugeordnet. Die modernen Kartoffelkultursorten stammen ursprünglich aus den Andenregionen Südamerikas zwischen Bolivien und Peru. Wilde Kartoffel wurde vor ca. 8000 Jahren domestiziert. Allerdings wurden die ersten Kultursorten von Kartoffel erst in den 1570ern nach Europa gebracht und haben sich dann von dort im späten 17. Jahrhundert weltweit verbreitet. Heute wird domestizierte Kartoffel weltweit, zwischen den Breitengraden 65°N bis 50°S, angebaut und kann selbst in Höhenlagen von bis zu 4000 m wachsen. Dies zeigt die immense Anpassungsfähigkeit zu verschiedensten Umweltbedingungen von Kartoffel. Trotz dieser besonderen Anpassungsfähigkeit weist Kartoffel eine hohe Sensitivität gegenüber Hitze und Trockenheit auf. Im Besonderen führen hohe Temperaturen (über 20°C) zu einer Reduktion der Knollenbildung und haben damit einen negativen Einfluss auf den Ertrag.
Aufgrund des sich stark ändernden Klimas, der immer weiterwachsenden Weltbevölkerung sowie der Notwendigkeit Lebensmittel selbst unter ungünstigen klimatischen Bedingungen anbauen zu müssen, arbeiten wir in drei Projekten im Biocomputing Lab, mit Hilfe nationaler und internationaler Partner daran Cassava und Kartoffel zu verbessern. Für Details zu den einzelnen Projekten können Sie sich gerne an den jeweiligen Doktoranden oder unseren Arbeitsgruppenleiter (Stephan Reinert) wenden.
- Verbesserung von Ertragsparametern von Cassava (Sindy Gutschker)
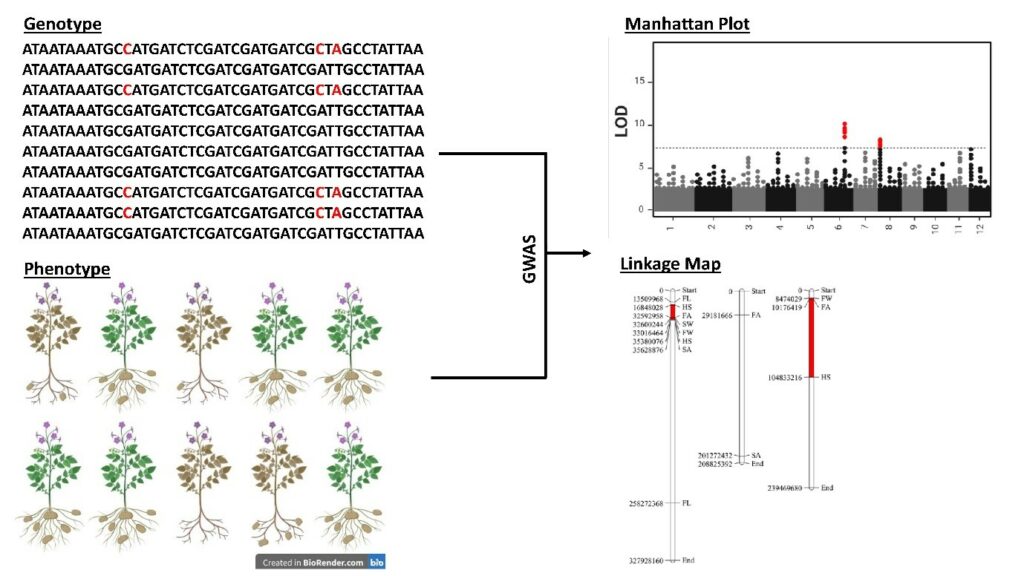
Unglücklicherweise ist die konventionelle Züchtung von Cassava sehr zeitintensiv. Außerdem braucht es oft viele Jahre, bis eine verbesserte Cassavasorte marktreif ist. Aus diesem Grund arbeiten wir, als bioinformatischer Projektpartner des CASS-Projekts, daran, den konventionellen Prozess der Cassavazüchtung unter der Verwendung verschiedenster bioinformatischer Methoden zu beschleunigen. Hierfür verwenden wir modernste „Omics“-Technologien wie Transkriptomik, Genomik, expressionsbasierte genomeweite Assoziationsstudien (eGWAS), transkriptomweite Assoziationsstudien (TWAS) sowie vergleichende Analysen (Clustering-, Assoziations-, Regressions- und Dimensionsreduktions-Methoden). Im Rahmen dieser Analysen sind wir besonders an agronomischen, agro-morphologischen und biotischen/abiotischen Resistenzmerkmalen interessiert.
- Verbesserung der Hitzetoleranz von Kartoffel (Alexander Kaier)
In unserm Kartoffelprojekt sind wir besonders an den agronomischen Merkmalen Stärkegehalt in Knollen und Ertrag unter Hitzestress interessiert. Unter der Zuhilfenahme von GWAS mit unseren genotypischen und phänotypischen Informationen von den über 250 Kartoffelsorten sind wir in der Lage wichtige potenzielle Kandidatengene zu detektieren, die eine Verbesserung der Hitzetoleranz in Kartoffel erzeugen. Durch die Verwendung dieser potenziellen Kandidatengene in Transkriptom- und Genstrukturanalysen versuchen wir die zugrundeliegenden genetischen Mechanismen und Faktoren der Hitzetoleranz in Kartoffel besser zu verstehen und können diese dann Kartoffelzüchtern als neue Ziele für ihre Zuchtprogramme präsentieren.
- Epigenetik der Hitzetoleranz bei Kartoffeln (Darren Yeo)
Im Laufe des letzten Jahrzehnts konnte festgestellt werden, dass wichtige agronomische Merkmale wie der Ertrag stark durch epigenetische Faktoren reguliert werden, besonders unter ungünstigen, klimatischen Bedingungen. Unter Epigenetik verstehen wir die Regulation der Expression von Genen mit Hilfe von Modifikationen des Genoms, ohne dabei die zugrundeliegende Genomsequenz zu verändern. Eine der möglichen Modifikationen ist die Methylierung und Demethylierung des Cystosin. Die Methylierungsmuster können zwischen verschiedenen Genotypen, Individuen oder sogar verschiedenen Geweben innerhalb eines Individuums stark variieren und korrelieren häufig mit Veränderungen in der Umgebung, z. B. bei Hitzestress. Aus diesem Grund ist unser Ziel, die Methylierungsmuster in zwei Kartoffelgenotypen, welche unterschiedlich stark auf Hitze reagieren, zu untersuchen. Mittels dieses Vergleiches wollen wir unterschiedlich methylierte Genomregionen identifizieren, welche möglicherweise zu einer Veränderung der Genexpression und somit Hitzetoleranz führen.

